Le mécanisme de la protéine UHRF1 mis en lumière
Des chercheurs du Laboratoire de bioimagerie et pathologies (LBP – CNRS/Unistra), en collaboration avec des laboratoires espagnols, américains, et italiens ont mis en lumière le mécanisme de la protéine UHRF1 dans la transmission fidèle des profils de méthylation de l’ADN lors de la division cellulaire. Un mécanisme qui représente une cible thérapeutique dans le traitement contre le cancer.
Différents mécanismes existent pour contrôler l’expression du génome parmi lesquels l’épigénétique, qui joue un rôle clé pour préserver l’identité des différents types de cellules
, explique en préambule Yves Mély, membre du laboratoire.
Dans l’épigénétique, un rôle particulièrement important est joué par la méthylation d’un ensemble de cytosines présentes dans des régions régulatrices de l'expression des gènes. Cet ensemble de cytosines méthylées du génome constitue le profil de méthylation, qui est spécifique à chaque type cellulaire. Lors de la division de la cellule, un brin ADN de la cellule mère est conservé et sert de matrice pour la fabrication du brin complémentaire. Au cours de cette copie, le brin complémentaire n’est pas méthylé.
La protéine UHRF1 intervient à ce stade pour assurer la transmission fidèle du profil de méthylation de l’ADN à la nouvelle cellule. Son rôle est de lire l’ADN afin de reconnaître les cytosines méthylées du brin de la cellule mère et recruter une seconde protéine : la méthyltransférase DNMT1 pour recopier cette marque épigénétique sur le brin complémentaire.
Deux étapes identiques
Pour étudier plus précisément la lecture de la séquence par UHRF1, les chercheurs ont utilisé des sondes fluorescentes mais aussi des mesures cinétiques rapides, sans oublier la modélisation moléculaire et des calculs de mécanique quantique.
Ces techniques leur ont permis de montrer que contrairement à ce qui était admis, UHRF1 lit non seulement les cytosines méthylées de l’ADN mais aussi celles non méthylées par un mécanisme de basculement de ces cytosines de l’ADN vers la protéine. La lecture par UHRF1 des deux types de cytosine est identique mais après cette étape de lecture, les cytosines non méthylées rebasculent rapidement dans l’ADN et la protéine poursuit sa lecture, alors que quand il s’agit d’une cytosine méthylée, UHRF1 se réorganise et recrute DNMT1.
Une cible thérapeutique
Cette découverte apporte un éclairage sur la façon dont UHRF1 distingue une cytosine méthylée d’une non méthylée, un mécanisme clé dans la mémoire épigénétique et la compréhension de certaines pathologies liées aux dérégulations de la méthylation de l’ADN comme les cancers.
Dans un cancer, les profils de méthylation sont altérés et UHRF1 est systématiquement surexprimée, contrairement à une cellule normale, où cette surexpression ne survient que lors de la division cellulaire.
Dans une autre publication à paraitre, les chercheurs ont identifié deux molécules qui bloquent la lecture des cytosines méthylées et empêchent le recrutement de DNMT1, provoquant la mort des cellules cancéreuses sans impacter les cellules saines.
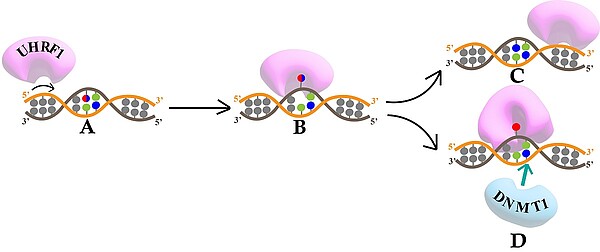
Le mécanisme de la protéine UHRF1 dans la réplication du profil de méthylation
Lors de la division cellulaire, le profil de méthylation du brin de la cellule mère (gris) doit être copié sur le brin complémentaire nouvellement synthétisé (orange). (A) Lecture de l’ADN par UHRF1. (B) Reconnaissance et basculement de la cytosine (bleu) ou de la méthylcytosine (rouge). (C) Lorsque la base basculée est une cytosine, cette dernière retourne rapidement dans l’ADN et UHRF1 poursuit sa lecture. (D) Quand la base basculée est une méthylcytosine, la protéine change de conformation et recrute la DNMT1 pour méthyler la cytosine du brin opposé. Les points verts désignent des guanines.